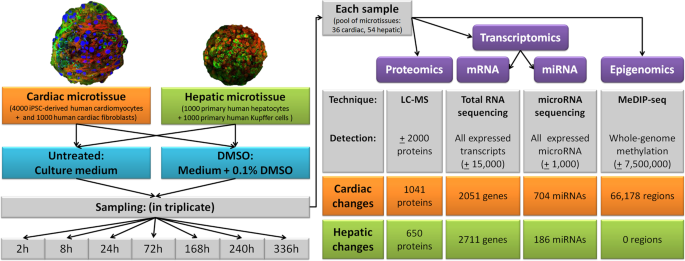
Menschliche 3D-Mikrogewebe (MTs) eines reifenden Herzmodells und eines reifen Lebermodells wurden zwei Wochen lang einem Kulturmedium mit oder ohne 0,1 % DMSO ausgesetzt, wobei die Probenentnahmezeitpunkte bei 2, 8, 72, 168, 240 und 336 Stunden lagen. Das Proteom (ungefähr 2.000 gemessene Proteine), das vollständige Transkriptom (einschließlich miRNAs) und die Methylierung des gesamten Genoms wurden an Material gemessen, das aus derselben Probe gewonnen wurde. Abbildung
1 enthält eine grafische Übersicht des Versuchsdesigns. Um einen ersten Überblick über DMSO-induzierte Cross-Omics-Effekte zu erhalten, werden die Mengen der unterschiedlich veränderten Einheiten (alle korrigiert für Mehrfachtests mit FDR <0,05) für jede Plattform zusammengefasst. Die Anzahl der unterschiedlich veränderten Einheiten unterschied sich zwischen den Gewebetypen, wobei Herzproben einen größeren Effekt von DMSO zeigten als Leberproben, mit Ausnahme von mRNAs. Dieser Unterschied ist besonders bei miRNAs und Genommethylierung auffällig. Da die Proteomikdaten aufgrund ihres partiellen Charakters am wenigsten aussagekräftig waren, wurden diese Ergebnisse in die ergänzenden Daten aufgenommen. Darüber hinaus zeigt die Hauptkomponentenanalyse (PCA) unter Verwendung von Durchschnittswerten aus dreifachen Proben für jede Plattform (Abb.
2 und ergänzende Daten) klare Unterschiede zwischen 0,1 % DMSO ausgesetzten (DMSO) und unbehandelten (UNTR) Proben, mit Ausnahme der Methylierung in Leber-MTs.